
作製した合金の結晶構造を調べるため、XRDを使う理系大学生はそれなりに多いと思います。
生データの扱いや比較するためのデータに困っている人におすすめなのが「PDIndexer」です。
今回はマニュアルもありますが少し使い方に癖のあるツールについて、サクッと解説します!
マニュアル:file:///C:/Users/hagi1/Downloads/PDIndexerManual(ja)%20(1).pdf
ダウンロード
本ツール開発者は京大大学院を卒業後、現在は大阪公立大学に所属している瀬戸 雄介准教授です。
まず彼のホームページ「Seto’s Page」アクセスして、「実行環境」の項目に進んでください。
.NET 6.0 Desktop Runtimeというツールを動かすための環境をMicroSoftからダウンロードします。
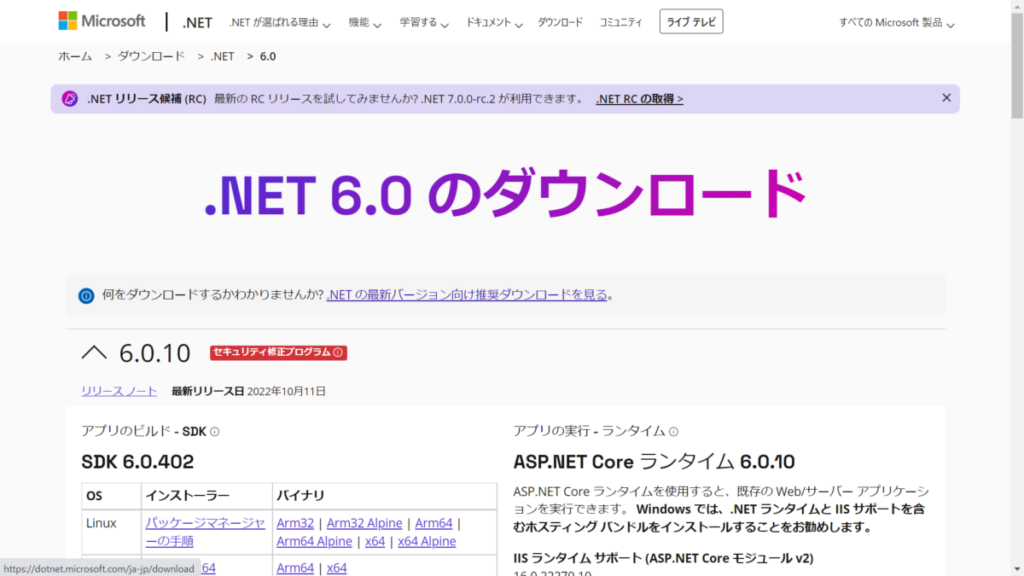
「.NETの最新バージョン向け推奨ダウンロードを見る。」をクリックして最新版を表示。
おそらくですが、Windowsで動かすことを前提としているので少なくともMacは無理そう。
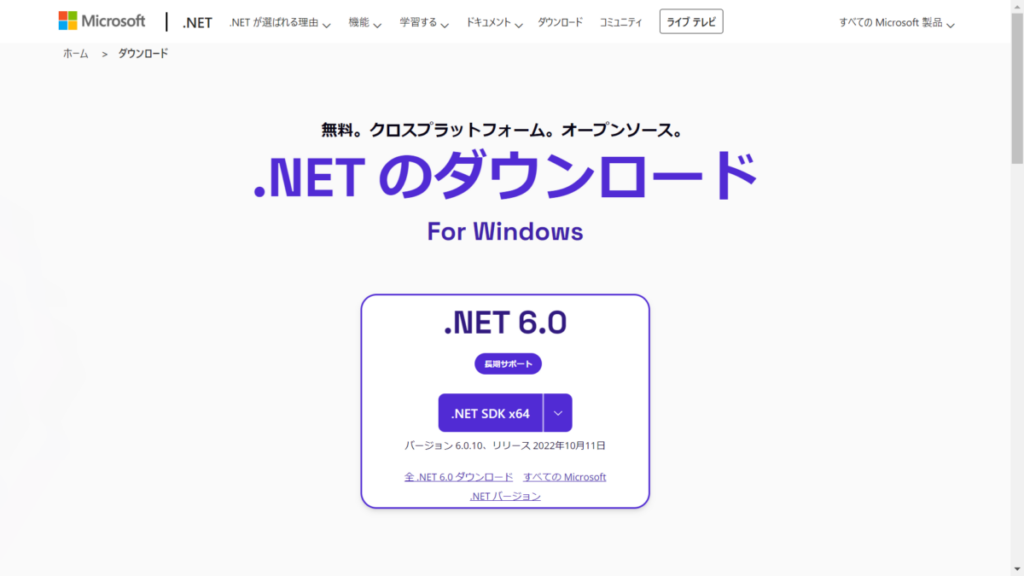
そのまま「.NET SDK x64」をクリックするとおそらく警告が出ますが、無視して実行。
ダウンロードが開始され、終了後開いたらファイルを保存する場所を選択して保存します。
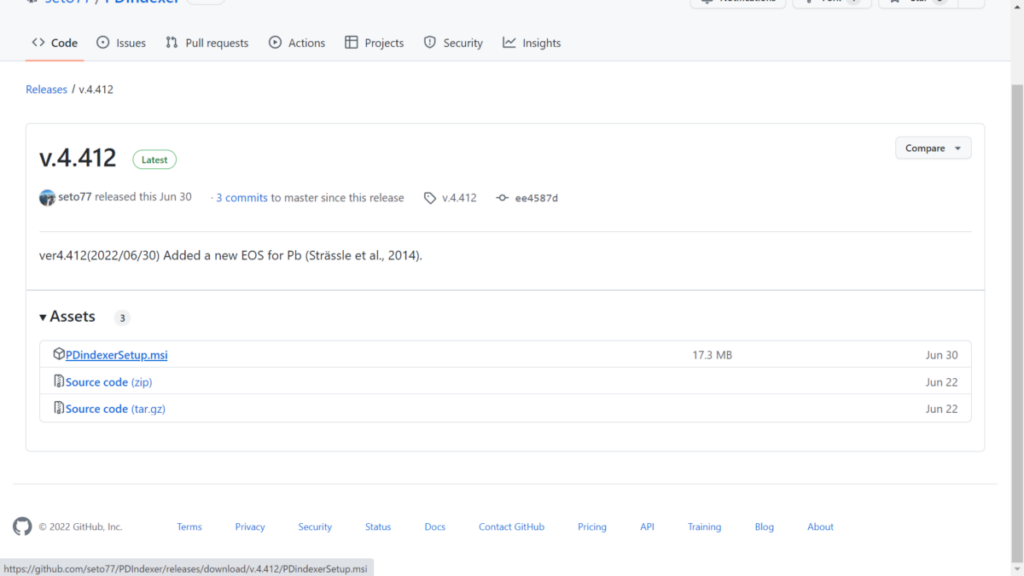
次に元のページから「Install」の項目にあるURLをクリックします(警告は無視)。
普通に一番上の「PDindexerSetup.msi」をダウンロードすれば開くことができます。
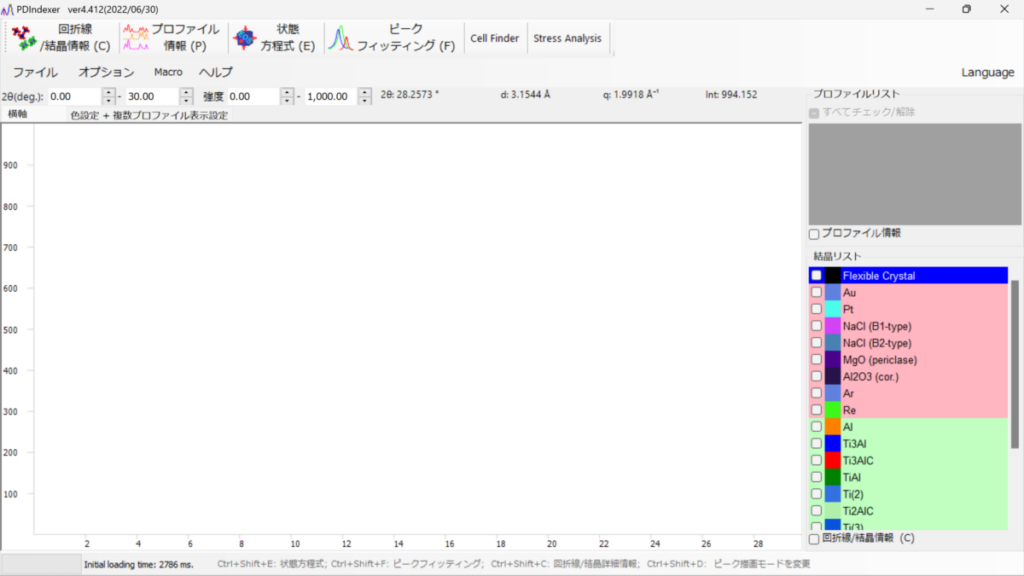
表示されたアイコンを立ち上げるとこのような画面が表示されるはず(緑色の部分はないかも)
次はここに保存しておくための、比較したい物質のデータをダウンロードしに行きます。

NIMS 物質・材料データベース (MatNavi)にアクセスし、メールアドレスで会員登録を行います。
登録したらページの下部に移動し、データベースの中の「Atom Work」を選択しログイン。
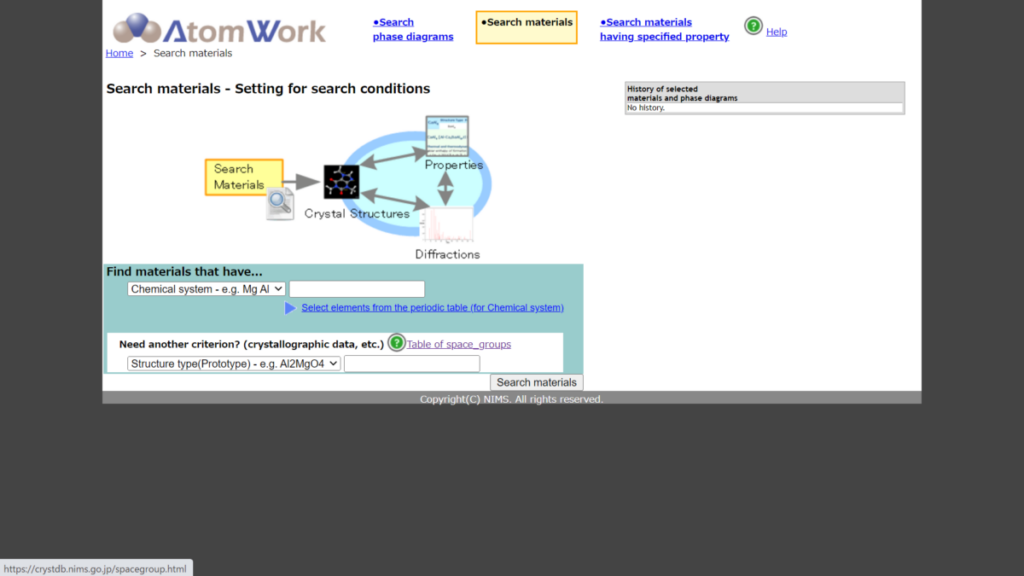
データベース検索画面のうち、青い文字の「Select elements from the periodic table」をクリック。
周期表の中から該当する元素を選択し、検索ボタンを押すと検索結果が表示されます。
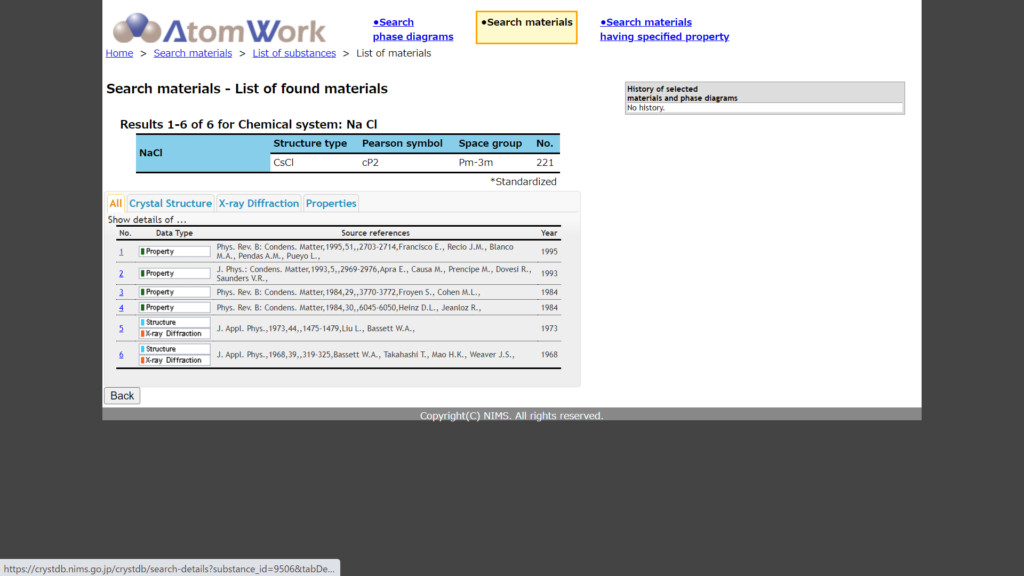
複数ある候補のうちから選択すると、該当するデータが新しい順に表示されます。
ここで選ぶのは水色とオレンジの項目があるもの(最低オレンジだけでもいいが少ない)
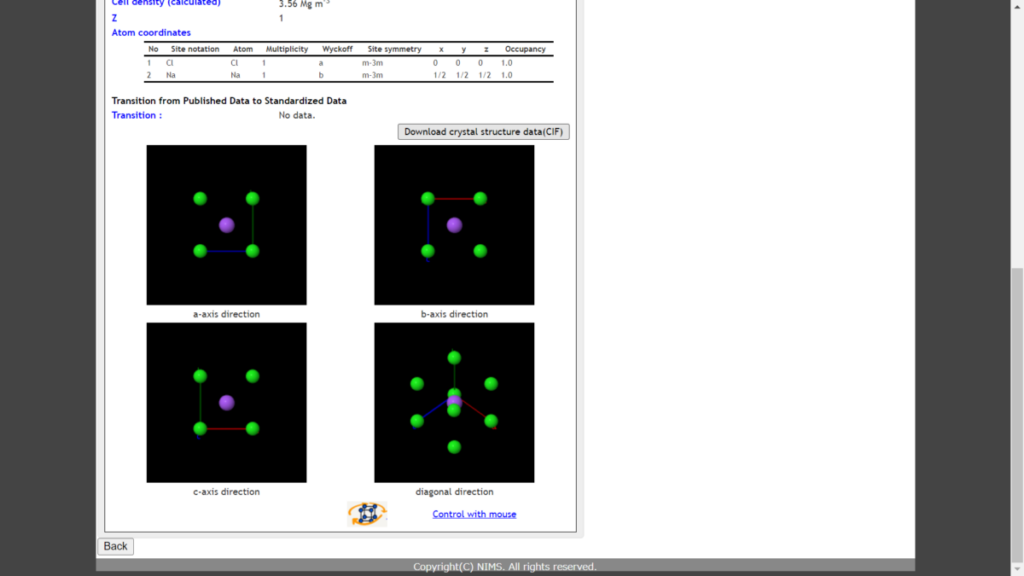
下に行くと結晶構造の画像が出てきます(Control with mouseで回転させることも可能)
XRDで用いるデータはこの画像の右上にある「Download crystal structure data(CIF)」です。
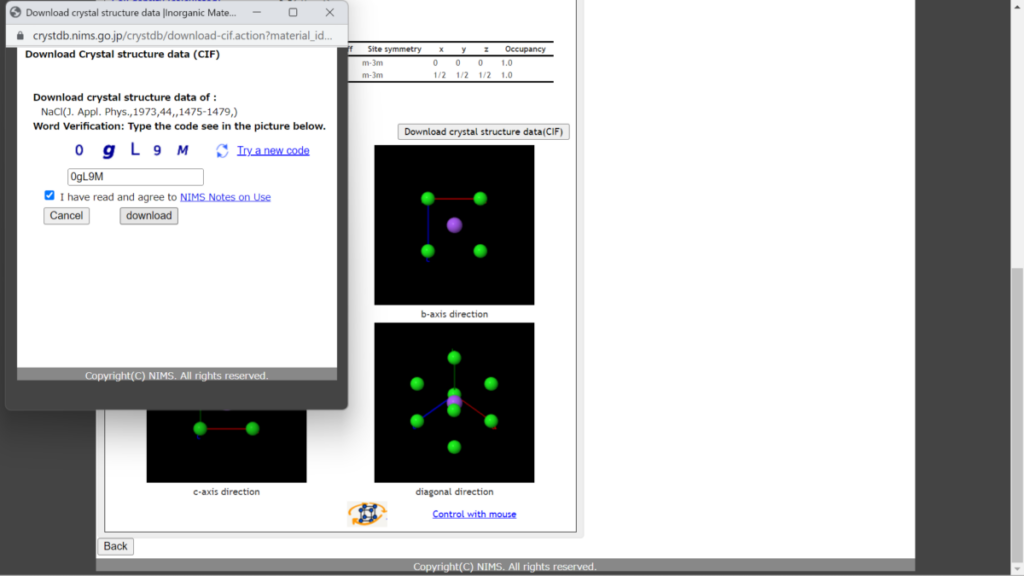
表示された英数字を入力し、下の「I have read and agree to NIMS Notes on Use」にチェック。
Downloadを押すと.cif形式で保存されます(1日に30件まで、名前は変更しておくといいかも)
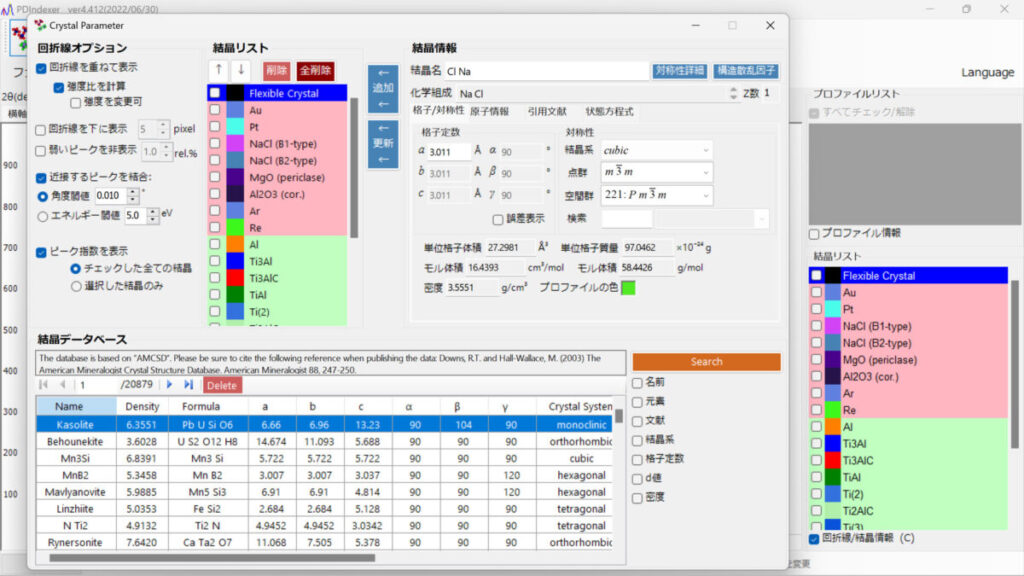
保存した.cifデータをPDIndexerにドラッグ&ドロップすると、新しいウィンドウが開きます。
もし変更するとしても結晶名くらい、画面中心の「追加」を押すと右下のデータベースに追加。
測定して得たデータも同じようにドラッグ&ドロップ、物質のデータと比較してみましょう。



